Équipe projet : Nathalie Aussenac-Gilles, Farah Bénamara, Catherine Comparot, Mouna Kamel, Elena Manishina et Cassia Trojahn.
Le projet RELTEX vise à utiliser les collections et les outils actuels ISTEX pour faire progresser la recherche en extraction de relations sémantiques à partir de textes en traitement automatique des langues et en représentation des connaissances.
L’identification de relations sémantiques en corpus contribue à l’extraction de connaissances et à la construction de ressources lexicales ou sémantiques (bases de connaissances) dans les domaines reflété par le corpus. L’identification d’une relation sémantique en corpus consiste à repérer deux termes, chacun désignant une entité (ou un ensemble d’entités), et à qualifier la relation exprimée dans le texte entre ces deux entités. Même pour un humain, cette tâche, non triviale, nécessite une compétence dans le domaine, et dépend de ce qui va être fait des relations. Il s’agit d’un processus subjectif, plusieurs personnes pouvant proposer divers ensembles de relations à partir d’un même extrait de texte. Cette tâche est encore plus critique dans les documents scientifiques très spécialisés dont l’interprétation requiert une grande expertise.
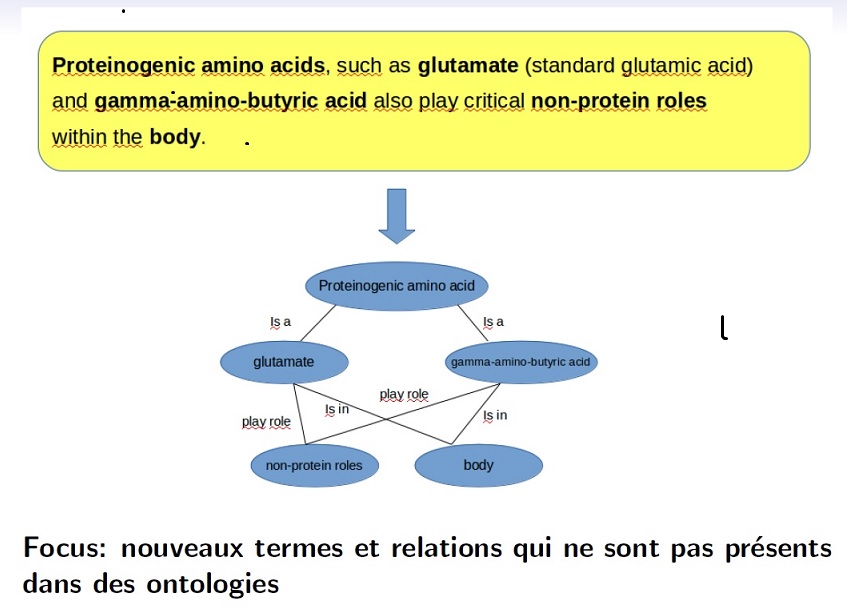
Par exemple, de la phrase :
\emph{Proteinogenic amino acids, such as glutamate (standard glutamic acid) and gamma-amino-butyric acid also play critical non-protein roles within the body}, les relations suivantes peuvent être extraites : \textit{glutamate} is\_a \textit{proteinogenic amino acid}, \textit{gamma-amino-butyric acid} is\_a \textit{proteinogenic amino acid}, \textit{glutamate} is\_in \textit{body}, \textit{gamma-amino-butyric acid} is\_in \textit{body} et \textit{proteinogenic amino acid} plays\_role\_in \textit{body}. Mais aussi \textit{glutamate} is\_a \textit{standard glutamic acid} ou encore \textit{glutamate} is\_a \textit{acid}.\\
Dans ce cadre, notre équipe MELODI a développé depuis plusieurs années des compétences en matière d’extraction de relations sémantiques à partir de textes, en s’intéressant à différentes approches (linguistiques, statistiques), sur des corpus de natures différentes (encyclopédique, scientifique, journalistique, etc.), usant de structures à des niveaux différents.
Grâce au projet RELTEX, nous avons souhaité améliorer et valider ces méthodes et en évaluer la complémentarité. Nous avons prévu de mettre en \oe{}uvre des logiciels basés sur des approches différentes sur un corpus scientifique extrait de la collection ISTEX. Cette collection semble bien se prêter à ce genre d’étude a priori : elle propose des sous-collections spécialisées, dans plusieurs langues (français ou anglais) ; chaque article contient des formes variées de rédaction et d’expression des connaissances (texte linéaire, titres, définitions, tableaux, etc.) ; enfin, certains documents sont disponibles sous plusieurs formats (txt, pdf, XML, …).
Notre projet s’est organisé selon un processus classique d’étude en traitement automatique des langues : mise au point d’un corpus et identification de ses particularités, identification de l’état de l’art, élaboration d’hypothèses concernant le choix de méthodes (basées sur l’apprentissage automatique) et les caractéristiques du corpus à prendre en compte, implémentation de ce modèle, évaluation et comparaison avec d’autres approches.
Pratiquement, la mise au point d’un corpus et nos premières expérimentations nous ont permis de préciser et réviser nos objectifs initiaux. Alors que nous avions prévu de mettre l’accent sur la prise en compte de la mise en forme et d’éléments structurés dans les articles, nous avons réorienté notre travail pour tester, sur ces corpus spécialisés d’assez petite taille, des approches basées sur de l’apprentissage supervisé ou non supervisé, à base de réseaux de neurones, dont on connait la pertinence sur de très grandes collections générales. Nous avons sélectionné un corpus cohérent parmi tous les textes de la collection ISTEX, composé de tous les articles disponibles d’une même revue scientifique en anglais (Nature). Nous avons choisi une méthode de supervision distante, à l’aide d’une ressource sémantique adaptée (une ontologie bio-médicale), afin de s’affranchir du travail d’experts pour l’annotation d’exemples de relations en corpus. Nous avons implémenté un algorithme basé du les cartes de Kohonene (CRM) pur identifier des classes de triplets correspondant à des types de relations. Nous avons réalisé plusieurs expérimentations, en utilisant ou non un extracteur de termes pour trouver les arguments en relation, avec différents types de traits pour la dsecription des triplets. NOus avons comparé cete approche à deux approches alternatives.
Les résultats partiels ont donné lieu à 2 publications :
- MANISHINA, M. KAMEL, C. TROJAHN, N. AUSSENAC-GILLES. Apprentissage non supervisé pour l’extraction de relations d’hyperonymie à partir de textes scientifiques. ACFAS2017 : Analyser la science : les bibliothèques numériques comme objet de recherche, Montréal (Canada), 08-09/05/17, Da Sylva, P. Cuxac (Eds.), 2017.
- MANISHINA, M. KAMEL, C. TROJAHN, N. AUSSENAC-GILLES. Unsupervised relation extraction from scientific texts using self-organizing maps. Atelier sur l’ Extraction et la Modélisation de Connaissances à partir de textes scientifiques, associé à PFIA2017, Caen (France), 03/07/2017, D. Buscaldi, V. Presutti (Eds.), AFIA, (en ligne), juin 2017.
Présentation lors du séminaire technique 2017


